- ·
Nanopore cDNA-PCR测序
- 产品介绍
- 案例解析
- 结果展示
- 送样建议
服务介绍
Nanopore cDNA-PCR测序是指基于牛津纳米孔公司(Oxford Nanopore Technologies,ONT)三代测序平台进行全长转录组测序,无需打断,可直接读取从5’端到3’端polyA尾的高质量单个RNA分子全长序列,准确辨别二代测序无法准确识别的可变剪接(AS)、融合基因、lncRNA及其靶基因,且可同时对基因和转录本进行定量分析。ONT全长转录组已广泛应用于生长发育、环境适应、免疫互作、突变表型、肿瘤的发生、临床诊断和药物研发等领域。
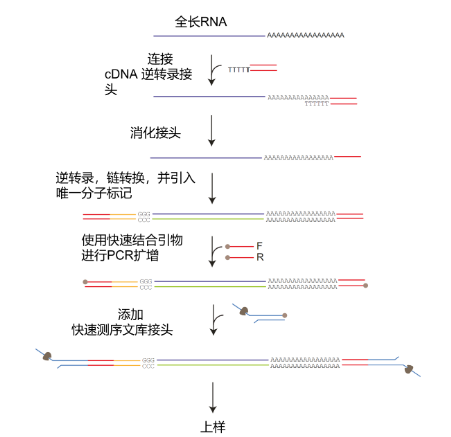
Nanopore cDNA-PCR测序流程图
测序方案
测序模式:Nanopore Sequencing
测序数据量:4Gb/样,or 6Gb/样
技术优势
1. Nanopre全长转录组测序无需打断RNA,可获得5’到3’全长转录本序列及其表达信息,对片段大小无偏好,直接检测电信号无需边合成边测序其GC偏好性远低于二代平台;
2. 由于无需拼接其在转录本层面的结构变异检测方面,比如可变剪接、融合基因、APA、新基因预测等具有绝对优势;
3. 除了可准确鉴别转录本结构变异,还可实现转录本(mRNA或polyA+ lncRNA)表达水平准确定量。
Nanopore long-read RNAseq reveals widespread transcriptional variation among the surface receptors of individual B cells
发表杂志:Nature Communications
影响因子:12.121
短reads RNAseq解析复杂isoform的能力有限,因为它无法测序RNA分子的全长cDNA拷贝。作者研究了使用长读取单分子Oxford Nanopore测序仪的RNAseq是否能够在不牺牲准确的基因表达定量的情况下,鉴定和定量复杂的isoform。在小鼠B1a细胞中鉴定了数千个未注释的转录起始和终止位点,以及数百个可变剪接事件,鉴定了在B1a细胞中表达的数百种基因,这些基因显示出多种复杂的isoform,包括几种B细胞特异性表面受体。本研究表明,可以在单细胞水平上识别和定量复杂的isoform。
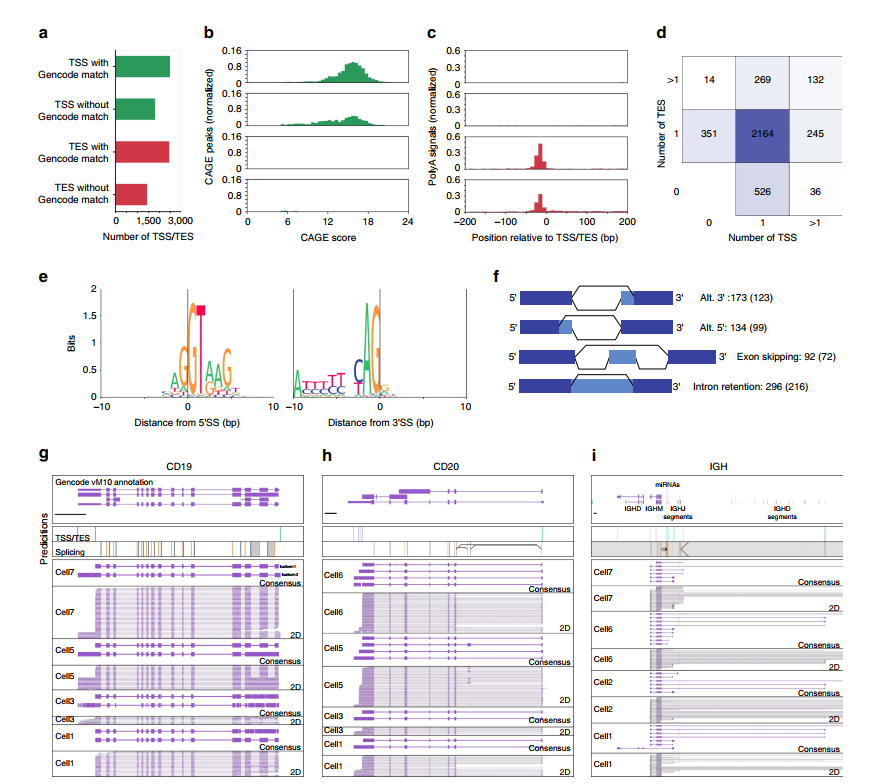
图1 ONT RNAseq数据分析确定小鼠B1a细胞的异构体特征
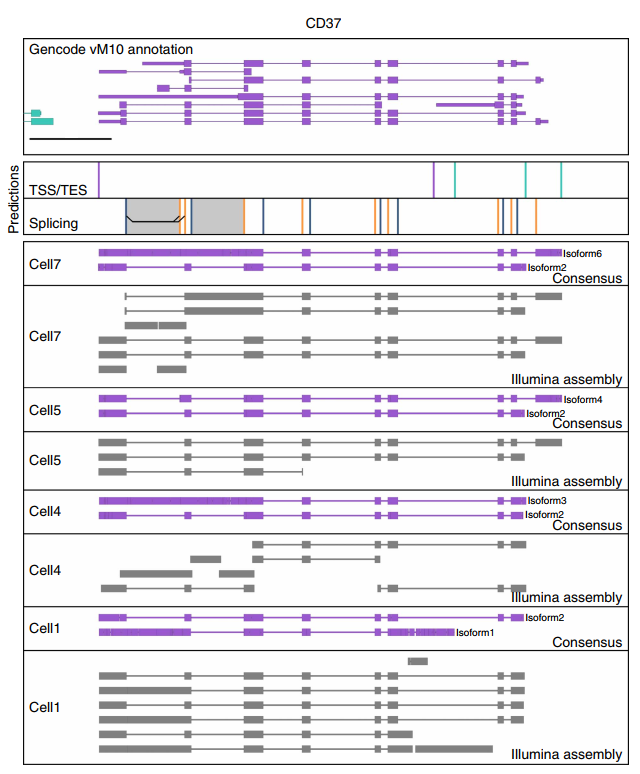
图2 揭示B细胞表面受体的异构体多样性
生信分析
ONT原始数据质控
比对结果质控检查
转录本定量
差异转录本鉴定
转录因子预测
功能注释
富集分析
蛋白质互作网络
可变剪接分析
融合转录本
新转录本发现
基因结构优化
Table. Nanopore cDNA-PCR Sequencing样本送样建议
送样类型
送样量
完整性(RIN值)
浓度
纯度
total RNA(组织、细胞、全血等)
≥2μg
≥8
≥100ng/μL
无DNA,蛋白/盐离子等污染,样本无色透明不粘稠
RNA带poly(A)尾
≥10ng
-
-
无DNA,蛋白/盐离子等污染,样本无色透明不粘稠
*更具体的送样方法请详询销售或技术支持
物种范围
人、小鼠、大鼠等哺乳动物,植物等其他物种详询销售或技术支持
填写需求描述给我们
工具快速咨询
400-8989-400
geneseed@geneseed.com.cn

 购物车
购物车




 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn