
- 首页
- >
- GENESEED
- >
吉赛ONT三代测序助力circRNA新研究!肖礼祖/唐七义/朱桦团队全面表征水痘带状疱疹病毒circRNA并揭示其生物学功能

作者:EP
校稿:杨少敏
水痘带状疱疹病毒(Varicella-zoster virus, VZV) 是一种双链DNA病毒,属于α疱疹病毒家族;是引起水痘(Varicella)和带状疱疹(Herpes zoster, HZ)的病原体。VZV初次感染人体的症状为躯干、头部或面部出现水泡性瘙痒皮疹,称为水痘(Varicella)(1),常见于10岁以下儿童,发生率约为1.40亿(2, 3)。VZV可在神经节神经元中建立潜伏期,再激活的VZV大量复制并沿感觉神经纤维向所支配的皮节扩散引起水痘(Varicella)皮疹,称为带状疱疹(Herpes zoster,HZ)(4, 5)。VZV病毒基因组大小约为124kp,传统认为其大约编码71个开放阅读框(Open Reading Frame, ORFs)(6),即基因。在VZV激活阶段(即裂解性感染,Lytic infection),71个ORFs均可转录大量病毒RNA。但在潜伏期间,VZV基因组DNA为环状结构;且几乎所有基因均处于静息状态,仅在ORF61附近表达VZV潜伏相关转录本(VZV latency-associated transcript, VLT) (7)。
最近研究利用先进的测序技术,如第二代短读测序和牛津纳米孔技术(ONT)长读测序,揭示了VZV感染细胞中复杂的转录组,并鉴定了许多非编码RNAs的非规范ORF(8)。该转录组包含以前未发现的转录本、各种转录异构体和值得进一步研究的剪接事件。
环状RNA (circRNA)是通过反剪接共价结合单链RNA形成的。circRNA缺乏5’帽结构和3’poly(A)尾的独特环状结构,使其比线性RNAs更耐受核酸外切酶,如RNase R。传统的circRNA分析方法需要从用RNase R处理总RNA ,然后通过短读RNA-seq,从数据中识别出反向剪接(BSJs)。最新的测序技术,尤其是结合RNase R处理和滚环反转录的ONT长读测序技术,为深入识别全长circRNA提供了更可靠的工具。在多种DNA和RNA病毒感染的细胞中已鉴定出病毒环状RNA。
华中科技大学协和深圳医院(深圳市南山区人民医院)疼痛科是国家临床重点专科疼痛科,2023年获评复旦大学医院管理所2022年度专科综合排行榜全国综合排名第三名,同时该科室拥有深圳市慢性疼痛重点实验室。自2014年建立以来以解决带状疱疹性神经痛临床问题为目标,开展多维度不同层次的研究。带状疱疹神经痛该院疼痛科的神经病理性疼痛的核心病种,每年约250~300例HZ或PHN住院患者在此得到规范治疗。
2024年6月10日, Nature Communications(IF=16.6)在线发表由华中科技大学协和深圳医院,联合美国罗格斯大学新泽西医学院和美国霍华德大学的研究性论文“Identification and Characterization of Varicella Zoster Virus Circular RNA in Lytic Infection”。博士后杨少敏(该院),实验员曹迪(该院)和Dabbu Kumar Jaijyan(罗格斯大学)为共同第一作者;唐七义教授(霍华德大学),肖礼祖主任(该院)和朱桦教授(罗格斯大学)为共同通讯作者。该研究采用二代短读测序和三代ONT长读测序鉴定和表征了VZV感染的神经母细胞瘤细胞和VZV中的环状RNA,鉴定了一种源自VZV潜伏期相关转录本(VLT)的VZV circRNA,并命名为circVLTSlytic,研究结果表明circVLTSlytic赋予了VZV对阿昔洛韦(ACV)的耐药性,揭示了VZV逃避抗病毒治疗的潜在机制。

VZV感染期间宿主环状RNA的动态
研究采用荧光检测/RT-qPCR/WB检测病毒相关指标;通过深度circRNA-seq鉴定细胞circRNA,并存入circBank(吉赛生物生信部联合加拿大多伦多大学创建的公开circRNA专业数据库,网址:http://www.circbank.cn/)。利用DESeq2/GO/KEGG等生信分析,发现大多数细胞circRNA的产生不受VZV感染的影响,VZV调控的细胞DE-circRNAs亲本基因与细胞生长调控、染色体分离、纺锤体、GTPase调节活性、核苷-三磷酸酶调节活性信号通路、轴突导向甲状腺激素信号通路有关。研究选择了VZV调控的6个重要的细胞DE-circRNA,并进行了Sanger测序和qPCR验证。

图1. VZV感染期间细胞circRNA的概况。
VZV编码circRNA的算法鉴定和表征
研究采用BGISEQ短读测序和ONT长读测序全面鉴定了VZV编码的circRNA,结果表明病毒circRNA随VZV感染时间而增加;较长的circRNA表达水平较低。短读测序具有高通量的优势,CIRI-full重建了42,563个人类全长circRNA,和其它部分组装的circRNA;而长读测序鉴定出12,407个全长人类全长circRNA;短读测序鉴定出71个全长VZV circRNA,而长读测序鉴定出1358个全长VZV circRNA,这体现了ONT滚环长读序列检测病毒circRNA中更高的效率和可靠性。进一步分析发现,大部分的VZV和细胞circRNA都含有多个环状外显子;VZV circRNA平均长度比细胞的短;VZV circRNA表现出大约50%的链偏好;VZV circRNA有多种异构体,如与潜伏期相关转录本(VLTs)同源的circVLTSlytic。
研究中ONT circRNA长读测序(Nanopore circRNA全长测序)由吉赛生物提供。Nanopore circRNA全长测序是利用随机引物对RNase R富集后的circRNA进行滚环反转录扩增,采用纳米孔测序技术对circRNA的全长序列进行直接测序,并使用特定算法,实现对长测序片段中的circRNA序列进行识别和全长重构。上述研究对比了二代与三代ONT测序技术,二代测序通量更高,但三代ONT测序在针对丰度更低的VZV circRNA的检测更灵敏。
三代ONT测序技术在检测circRNA上具有极大优势:
1. 更强的检出率:相比二代测序,纳米孔测序可以将circRNA reads检测效率提高20倍以上。
2. 更高的灵敏度:可识别低丰度的circRNA,能够更敏感地捕获到非经典circRNA,i.e., intronic。
3. 更多更准确的应用领域:
1)准确识别可变剪切事件,无需拼接,一键生成;
2)直接识别癌症等重大疾病发生发展的重要生物标志物一一融合环状RNA(f-circRNA);
3)能够更敏感的识别研究热点一一线粒体环状RNA,确保您的研究不会落空。
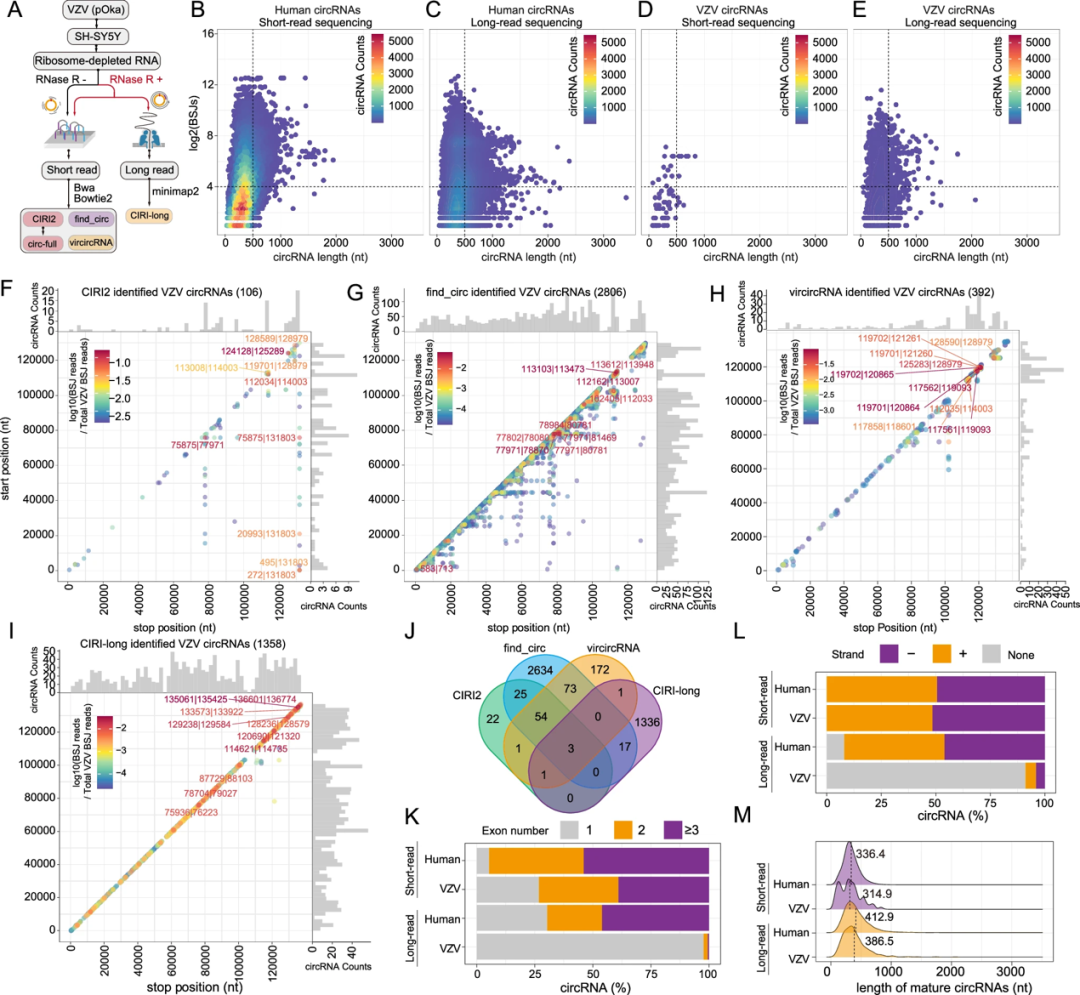
图2. VZV circRNA的鉴定及表征。
 图3. VZV circRNA和circVLTSlytic全长。
图3. VZV circRNA和circVLTSlytic全长。
VZV感染细胞中病毒circRNA的实验鉴定
研究通过RT-PCR、Sanger测序、RNase R敏感性试验和该团队发明的链式扩增FISH技术鉴定了测序得到表达量较高的VZV circRNA,结果表明这些病毒circRNA存在于VZV感染细胞中,circVLTSlytic含多个外显子,主要分布于VZV感染细胞的细胞核和细胞质。
研究中RNase R(GSPure®RNase R)由吉赛生物提供,RNase R是一种来源于大肠杆菌RNR超家族的3’-5’核糖核酸外切酶,可从3’-5’方向将RNA逐步切割成二核苷酸和三核苷酸。RNase R可消化几乎所有的线性RNA分子,但不易消化环形RNA、套索结构或3’突出末端少于7个核苷酸的双链RNA分子。RNase R是circRNA的鉴定和富集实验必备工具酶,可消化线性RNA以使环形RNA(circRNAs)或套索结构RNA(lariat RNA)得到富集。RNase R广泛应用于circRNA鉴定(如上述的RNase R敏感性试验)、富集(如上述的测序分析)、纯化(如circRNA制备)等。
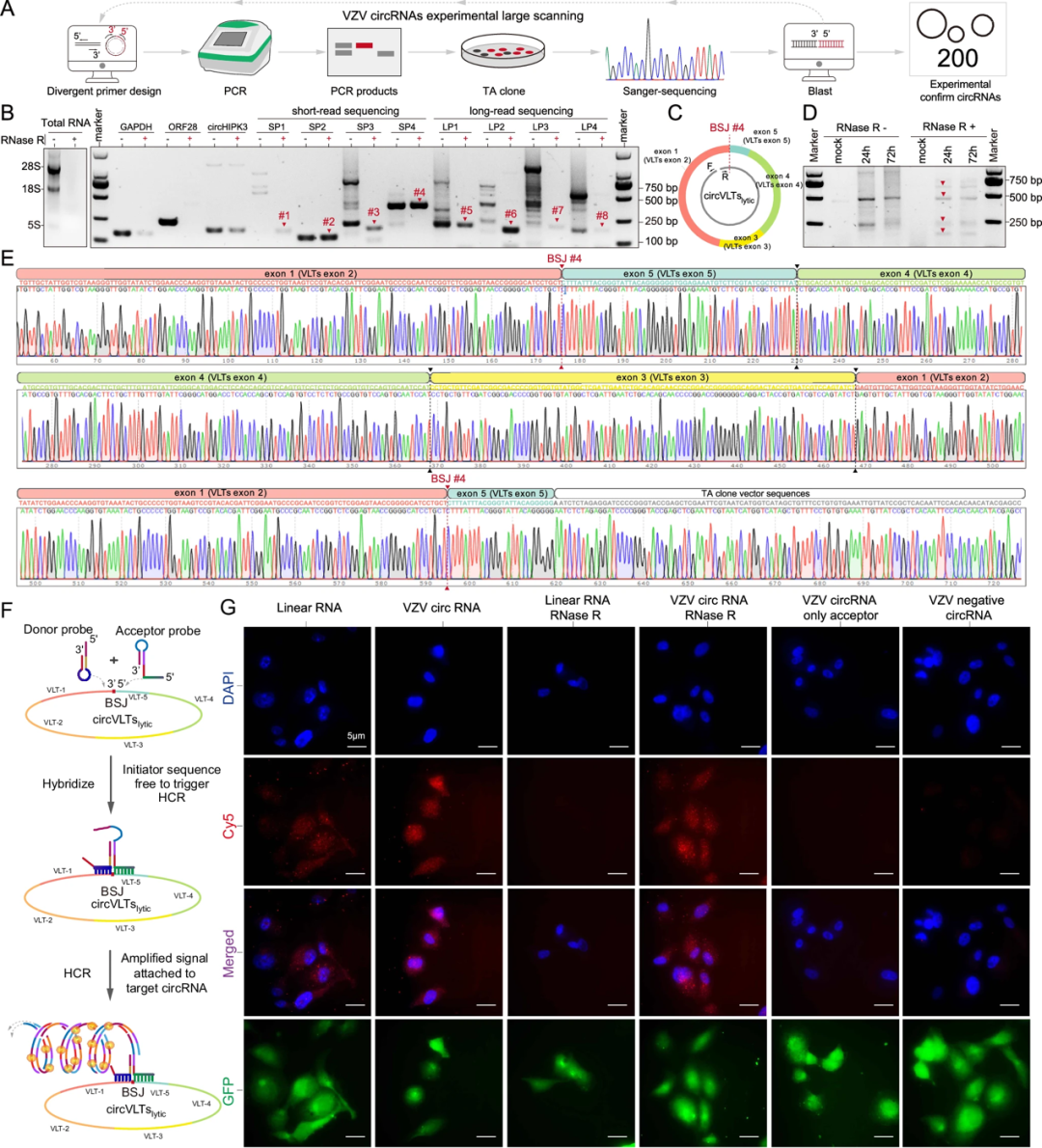
图4. VZV环状RNA的实验鉴定。
带状疱疹(HZ)患者组织和临床毒株中VZV circRNA的鉴定
研究收集6名被诊断为带状疱疹(HZ)患者的水泡液和分离的VZV毒株样本,经过RT-PCR和Sanger测序证明HZ患者水泡液和临床VZV毒株感染中都存在VZV circRNA,其中circVLTSlytic在以上两种样本中都存在。
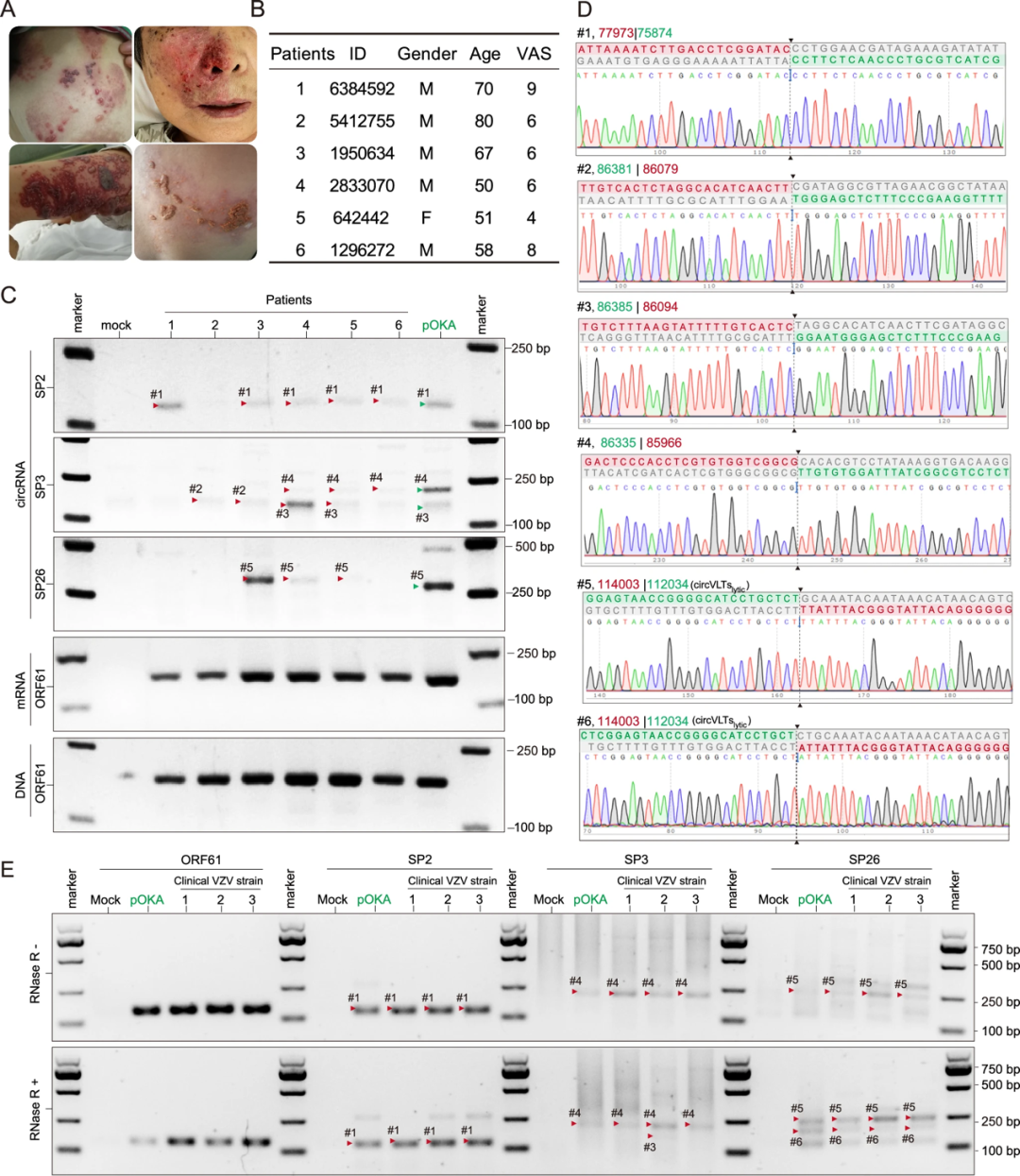
图5. HZ患者中VZV circRNA的鉴定。
circVLTs的DNA侧翼5’剪接供体是VZV circVLTSlytic形成的一个顺式元件
分析circVLTSlytic在基因组的位置发现,只有五号外显子不与VZV经典基因(ORF)发生重叠。因此,该团队利用细菌人工染色体(BAC)系统在pOka株(该病毒株已经插入了GFP和luciferase双报告基因)基因组DNA水平进行20个随机碱基替换突变,并产生pOka-M1(5’剪接供体下游突变)和pOka-M2(5’剪接供体上游突变)突变VZV株。结合RNA-seq和qPCR检测,结果表明,circVLTSlytic外显子5突变轻微抑制了RNA转录,且外显子5与外显子1有相互作用;DNA侧翼5’剪接供体附近序列对VZV circVLTSlytic剪接形成起关键作用,是VZV感染细胞后circVLTSlytic生物发生和表达所必须的顺式作用元件。
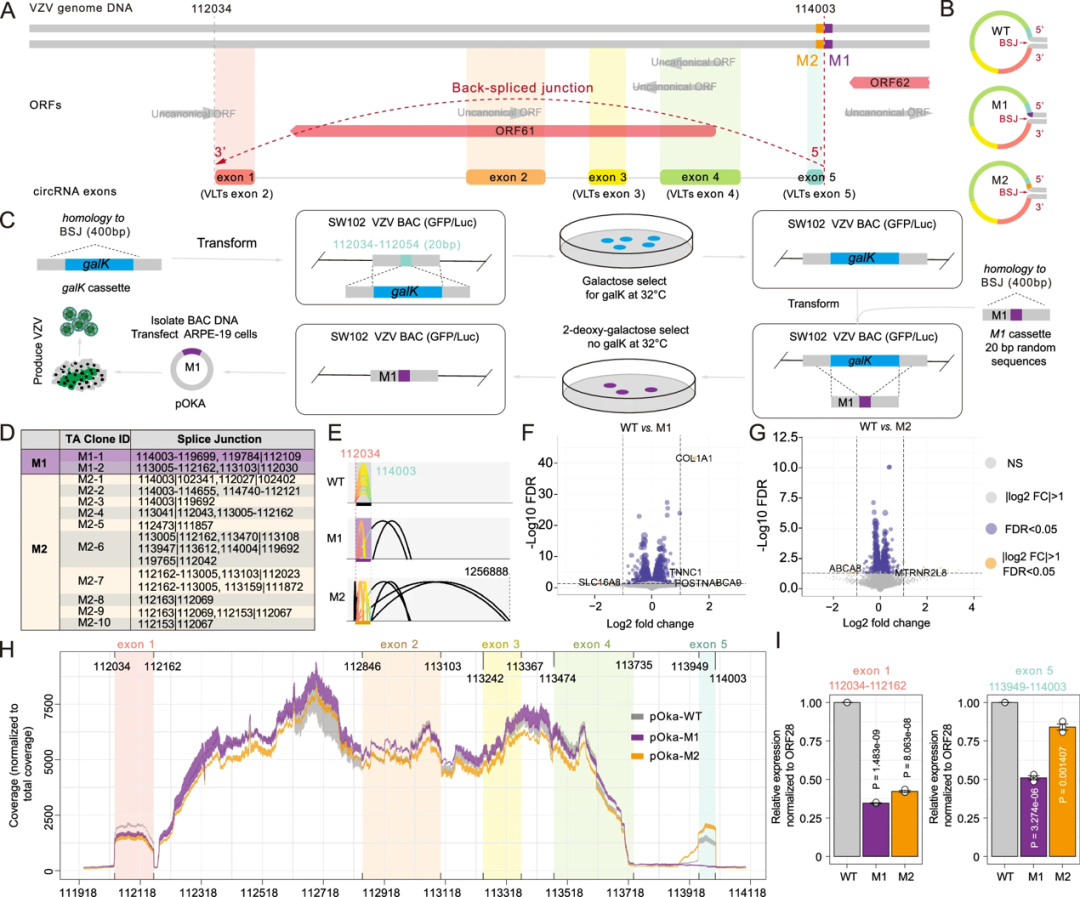
图6. circVLTSlytic基因组水平突变
circVLTSlytic外显子5的突变使VZV对阿昔洛韦更敏感
团队通过荧光素酶报告基因检测发现circVLTSlytic外显子5突变不影响VZV的生长;但用IFN-β、ACV或RDT三种抗病毒药物处理感染细胞给予VZV生长压力,发现pOka-M1对ACV的敏感性增加;补救实验(慢病毒载体过表达circVLT两种亚型)以及体外培养的人类皮肤、背根神经节以及人皮肤组织移植小鼠模型,并用ACV治疗,结果表明, circVLTSlytic是VZV复制的非必须分子,但其存在有助于VZV抵抗抗病毒药物的作用。进一步的研究表明,circVLTSlytic可能通过调控VZV基因的表达,从而响应抗病毒治疗。
研究中circRNA慢病毒过表达载体(pLCDH-ciR)由吉赛生物提供,吉赛生物第五代circRNA过表达载体系列能满足普通真核表达、慢病毒包装和腺相关病毒(AAV)包装等多种用途。载体均携带优化过的侧翼成环框架,含有精心改造的Alu元件、QKI等RBP的结合位点,并使用全新设计的环化介导序列,能保证插入的circRNA准确高效环化。吉赛生物第五代circRNA过表达载体环化具有更高的准确性、稳定性、通用性、可移植性。
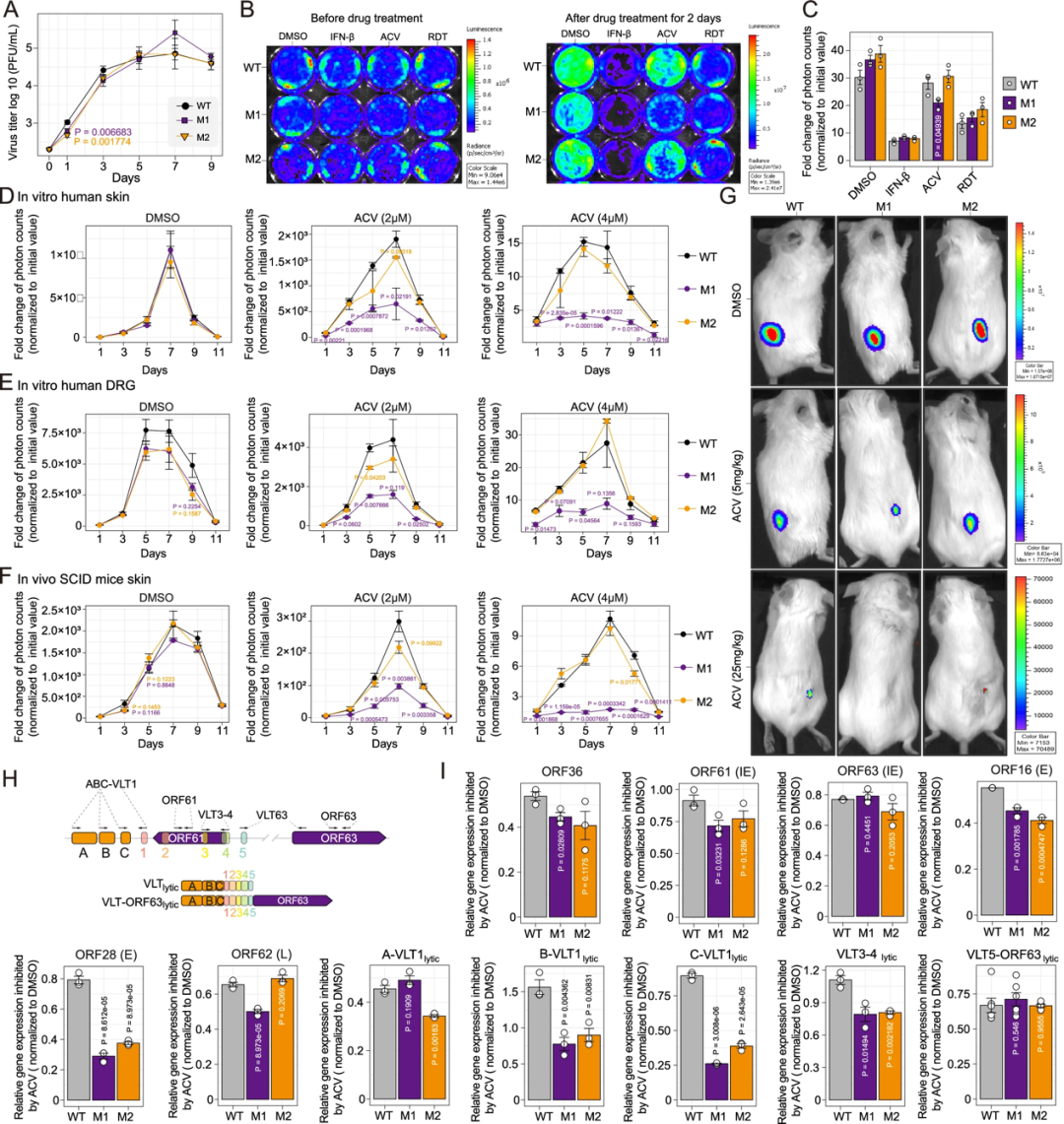
图7. circVLTSlytic外显子5的突变不会减弱VZV的复制,但会提高对ACV敏感性。
总结
该研究利用二代和三代测序技术以及生信分析鉴定及表征了VZV编码的circRNA,并在HZ患者感染细胞和组织中验证了这些circRNA的存在,报道一些新发现的VZV circRNA,还证明了circVLTSlytic 5'剪接供体下游序列的突变能增强VZV对阿昔洛韦敏感性,circVLTSlytic有利于VZV基因的表达,从而使VZV响应抗病毒治疗。这些结果证明VZV的感染过程中不止表达传统认知的71个ORFs,病毒来源的circRNA在VZV发病机制中可能发挥重要作用,揭示了circVLTSlytic在影响病毒耐药性中的潜在作用。
原文链接
https://www.nature.com/articles/s41467-024-49112-4
该文章相关代码和数据均已开源:https://github.com/ShaominYang/VZV_circRNA
参考文献
1. Gershon AA, Breuer J, Cohen JI, Cohrs RJ, Gershon MD, Gilden D, et al. Varicella zoster virus infection. Nat Rev Dis Primers. 2015;1:15016.
2. Thiele S, Borschewski A, Kuchler J, Bieberbach M, Voigt S, Ehlers B. Molecular analysis of varicella vaccines and varicella-zoster virus from vaccine-related skin lesions. Clin Vaccine Immunol. 2011;18(7):1058-66.
3. Varicella and herpes zoster vaccines: WHO position paper, June 2014--Recommendations. Vaccine. 2016;34(2):198-9.
4. 赵志奇. 带状疱疹痛:基础和临床概述. 中国疼痛医学杂志. 2014(6):369-75.
5. 带状疱疹后神经痛诊疗中国专家共识. 中国疼痛医学杂志. 2016;22(03):161-7.
6. Zhang Z, Selariu A, Warden C, Huang G, Huang Y, Zaccheus O, et al. Genome-wide mutagenesis reveals that ORF7 is a novel VZV skin-tropic factor. PLoS Pathog. 2010;6:e1000971.
7. Depledge DP, Ouwendijk WJD, Sadaoka T, Braspenning SE, Mori Y, Cohrs RJ, et al. A spliced latency-associated VZV transcript maps antisense to the viral transactivator gene 61. Nat Commun. 2018;9(1):1167.
8. Braspenning, S. E. et al. Decoding the Architecture of the VaricellaZoster Virus Transcriptome. mBio 11 (2020). https://doi.org/10.1128/mBio.01568-20

 购物车
购物车


 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn