
- 首页
- >
- GENESEED
- >
RNA机制研究秘籍:分子互作技术解析DNA-RNA-蛋白调控通路!
生信分析虽可描绘分子调控网络,却如雾里看花,唯有结合核心分子互作研究技术,方可精准解析DNA-RNA-蛋白的互作网络,实证RNA从表达调控到功能执行的完整通路。那么,核心分子互作技术如何贯穿应用于RNA机制研究全流程?
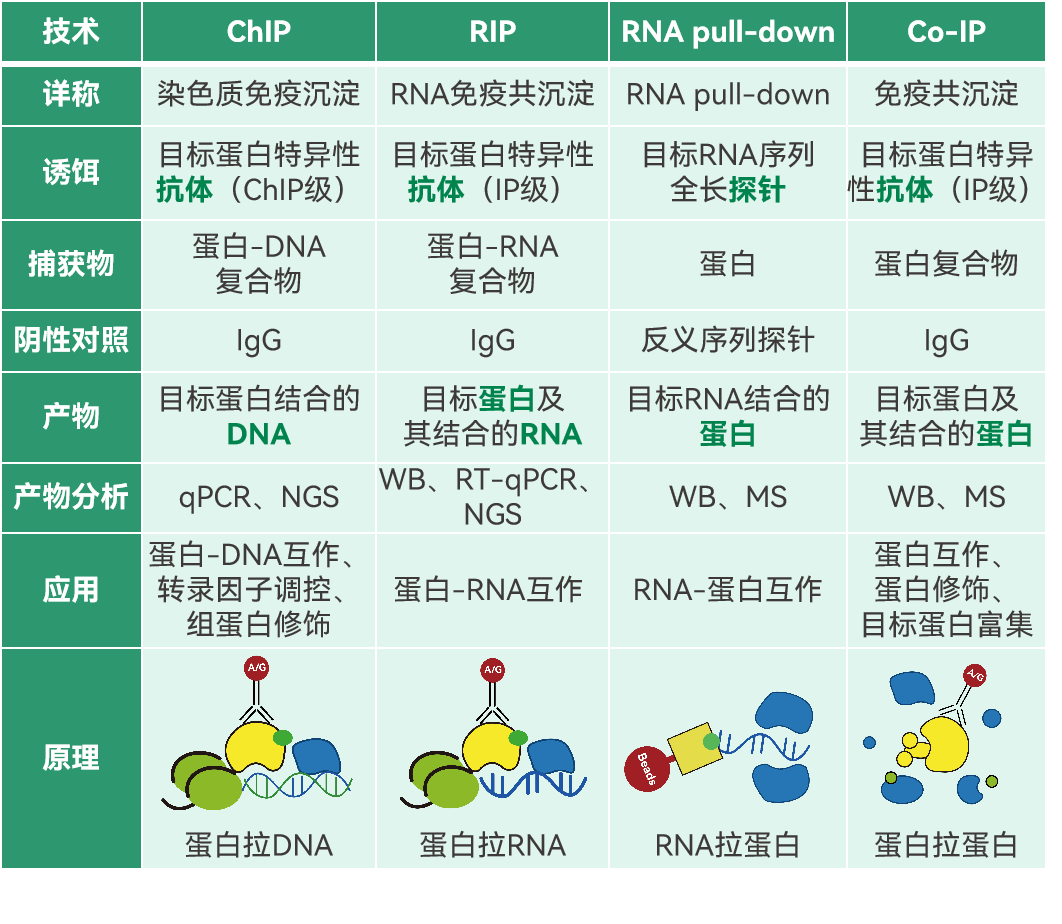
四大核心分子互作研究技术
追根溯源:目标RNA为何表达异常?
转录调控研究
ChIP-qPCR分析
● 转录调控因子与目标基因互作,分析结合动态变化的因素(如竞争性抑制剂的存在、转录调控因子突变、目标基因突变);
● 目标基因上转录复合物(如RNA聚合酶Ⅱ)的结合,发现新的调控机制,验证转录调控机制;
● 目标基因组蛋白修饰(如甲基化、乙酰化),揭示组蛋白修饰与基因表达的关联。
Co-IP-WB分析:转录调控因子与其它蛋白之间的互作;转录调控因子的蛋白修饰(如泛素化、磷酸化)。
转录后加工研究
RIP-qPCR:研究剪接相关蛋白与目标RNA前体的结合,分析剪接复合体的调控因素(如竞争性抑制剂的存在、剪接复合体突变)。
Co-IP:研究剪接复合体蛋白之间的互作。
RNA降解研究
RNA pulldown-WB/MS:筛选目标RNA结合的RNA降解/稳定性调控相关蛋白。
Co-IP:研究RNA降解相关蛋白与其它调控因子的互作。
RIP-qPCR/seq验证:
● 降解相关蛋白(如Ago2)结合目标RNA,同时分析其它ncRNA调控因子(如miRNA)对目标RNA降解的影响。
●m6A甲基化相关蛋白(如Writer、Eraser、Reader)结合目标RNA,研究RNA m6A甲基化的调控机制。
研究案例
主要研究内容:在胃癌(GC)中,转录因子EGR1促进TENM3-AS1转录,通过重编程脂肪酸代谢增强胃癌转移。[1]
TENM3-AS1转录调控研究
研究通过生信分析RNA-seq和数据库结果,预测与GC和TENM3-AS1表达相关的转录因子(TF)EGR1。ENCODE数据库中ChIP测序结果显示在H1和Ishikawa细胞中,EGR1与TENM 3-AS1启动子结合(图1A-B),使用JASPR预测结合位点(图1C)。anti-EGR1 ChIP-qPCR进一步验证EGR 1可重复结合TENM3-AS1的启动子的所有预测位点(图1D)。最后双荧光素酶报告基因实验验证了EGFR1可以促进TENM3-AS1的转录。
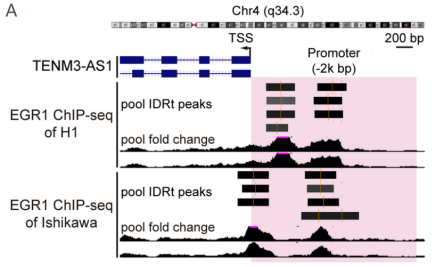
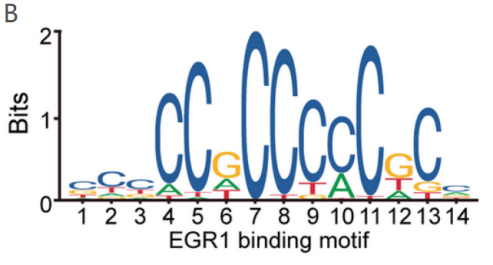
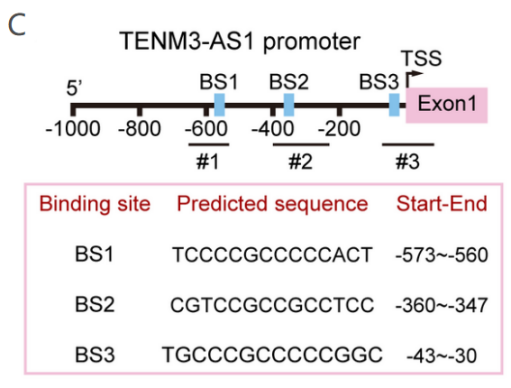
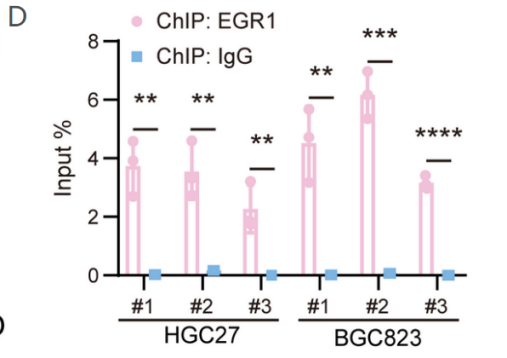
图1 anti-EGR1 ChIP验证EGR1与TENM 3-AS1启动子的结合。
顺藤摸瓜:目标RNA如何调控下游功能?
结合蛋白,影响蛋白功能或定位
RNA pulldown-MS:分析目标RNA结合蛋白谱,筛选功能表型变化相关的关键蛋白。
RIP-qCPR:验证关键蛋白与目标RNA的互作。
Co-IP-MS:分析RBP互作蛋白谱,研究RBP与目标RNA结合的影响因素;
Co-IP-WB:分析RBP修饰。
竞争性结合miRNA(ceRNA机制)
RNA pulldown(标签法)-seq:分析目标RNA结合的miRNA。
Anti-Ago2 RIP:分析关键蛋白Ago2结合的RNA,验证miRNA和目标RNA/靶RNA的动态结合关系。
结合mRNA,调控翻译
RNA pulldown(标签法)-seq:分析目标RNA结合的mRNA。
翻译多肽/蛋白
RNA pulldown-MS:分析目标RNA翻译调控蛋白。
Co-IP-WB/MS分析:
● 翻译调控蛋白的互作;
● 目标RNA编码多肽/蛋白的关键互作蛋白(信号通路、外泌体组装、细胞表型等相关蛋白),分析其对下游通路或表型相关蛋白的影响;
结合DNA或转录因子,调控转录
RNA pulldown-WB/MS:筛选目标RNA结合的转录调控因子;
RIP-qPCR:验证转录调控因子结合目标RNA;
Co-IP分析:转录调控因子互作蛋白;转录调控因子的蛋白修饰。
ChIP-qPCR分析:
● 目标RNA对转录因子结合靶DNA的影响。
● 目标RNA对RNA聚合酶结合靶DNA的影响,反映目标RNA调控靶基因转录。
研究案例
主要研究内容:circHAS2通过作为miR-1244的分子海绵,并结合USP10以促进p53核输出和降解,从而激活CCNE2促进细胞增殖,并使结直肠癌对安洛替尼的反应增敏。[2]
circHAS2的ceRNA机制验证
生信分析预测circHAS2与miR-1244的表达相关,anti-Ago2 RIP揭示与miR-1244 NC组相比,miR-1244 mimic组SW620细胞中,Ago2结合的circHAS2富集程度更高(图2)。然后双荧光素酶报告基因实验验证circHAS2通过ceRNA机制,抑制miR-1244,并上调CCNE2的表达。
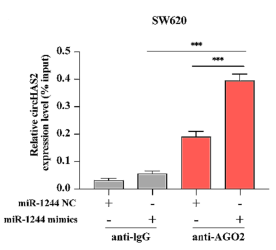
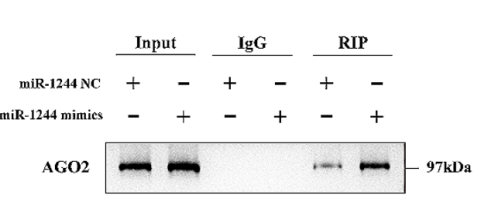
图2 用RIP法检测特定处理的SW620细胞中circHAS2和Ago2的相对表达。
circHAS2调控转录机制验证
circHAS2 RNA pulldown-MS分析显示在SW620细胞中,circHAS2可能结合泛素特异性肽酶10(USP10)。circHAS2 RNA pulldown-WB(图3A-C)和anti-USP10 RIP-qPCR(图3D),进一步验证了USP10和circHAS2的直接相互作用。
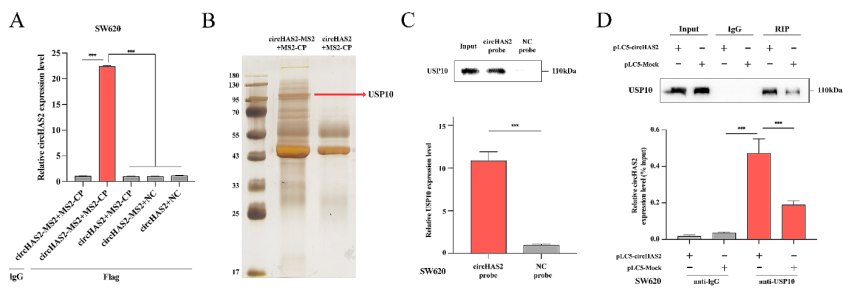
图3 circHAS2与USP10蛋白相互作用验证。
前人研究显示USP10可结合p53可相互作用,因此研究利用anti-USP10和anti-p53 Co-IP-WB验证了USP10与p53的结合(图4左);anti-USP10 Co-IP-WB研究显示过表达circHAS2,减弱了USP10与p53的结合(图4右)。
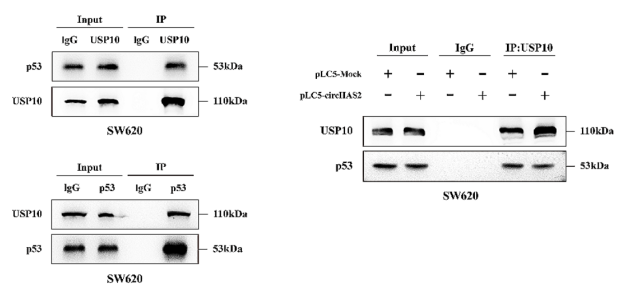
图4 Co-IP验证p53和USP10的结合,以及circHAS2对USP10和p53结合的影响。
在转染Flag-USP10、circHAS2或HA-Ub及MG132处理的SW620细胞中,anti-HA Co-IP-WB证实了circHAS2过表达促进p53蛋白泛素化和降解,该过程这可由USP10逆转。
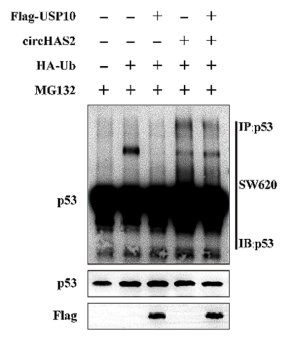
图5 Co-IP验证CircHAS2与USP10相互作用调节p53泛素化和表达水平。
此外,Co-IP-WB分析SW620细胞核与细胞质组分中p53的泛素化水平,验证circHAS2过表达增加了细胞质中的p53泛素化(图6)。最后验证了circHAS2促进p53的核输出,使其在细胞中泛素化和降解,从而下调p53靶基因p21的表达并上调CCNE2的表达。
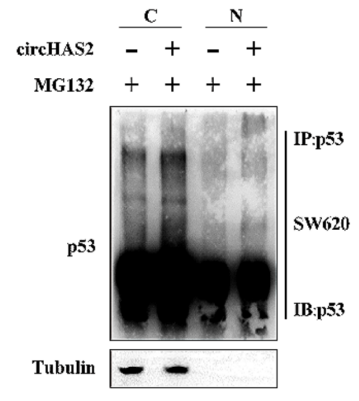
图6 Co-IP实验证实circHAS2上调细胞质中p53泛素化。
工欲善其事,必先利其器
吉赛生物匠心打造PureBinding®系列ChIP、RIPRNA pull-down、Co-IP四款分子互作研究核心试剂盒和技术服务,“经典百搭款”灵活适配多种分子机制研究场景,大规格试剂盒也不愁用还更划算!手握这四款研究利器,助您高效解密RNA调控机制,系统揭示RNA作用网络,加速科研突破,成果水到渠成!
技术 | PureBinding®产品名称 | 产品货号 | 规格 |
ChIP | P0301 | 12 rxns | |
P0302 | 24 rxns | ||
RIP | P0101 | 12 rxns | |
P0102 | 24 rxns | ||
RNA pull-down(探针法) | P0201 | 12 rxns | |
P0202 | 24 rxns | ||
Co-IP | P0401 | 12 rxns | |
P0402 | 24 rxns |
参考文献
[1]Tang Y, et al. The EGR1-mediated lncRNA TENM3-AS1 potentiates gastric cancer metastasis via reprogramming fatty acid metabolism. Mol Cancer. 2025;24(1):165.
[2]Li H, et al. CircHAS2 activates CCNE2 to promote cell proliferation and sensitizes the response of colorectal cancer to anlotinib. Mol Cancer. 2024;23(1):59.

 购物车
购物车


 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn