
- 首页
- >
- GENESEED
- >
科研辟谣|Polysome Profiling不就是离心甩一甩?这4个真相你必须知道!
在中心法则的研究圈里,有一个技术虽然诞生于上世纪60年代,却依然被奉为翻译研究的“金标准”。它就是Polysome Profiling(多聚核糖体图谱分析)。
很多人对它的印象还停留在“古老”、“麻烦”甚至“没必要”上。今天,我们就用4个问答,带你重新认识这个能帮你“拿捏”高分文章的关键技术。
转录组测序这么发达,为什么还要做Polysome?
因为“听到的”不等于“做到的”。
转录组告诉你细胞“想”做什么(mRNA水平),而翻译组告诉你细胞“正在”做什么(蛋白合成水平)。
研究表明,转录组和蛋白组的变化往往不一致。特别是在以下场景中,Polysome Profiling是无可替代的:
应激反应(Stress):缺氧、药物刺激时,细胞来不及转录新RNA,而是直接调控现有RNA的翻译效率。
病理机制(Pathology):致病蛋白的过量表达,往往源于翻译起始的异常激活,而非转录增加。
只有通过Polysome Profiling计算翻译效率(TE),你才能解释为什么mRNA没变,蛋白却变了。
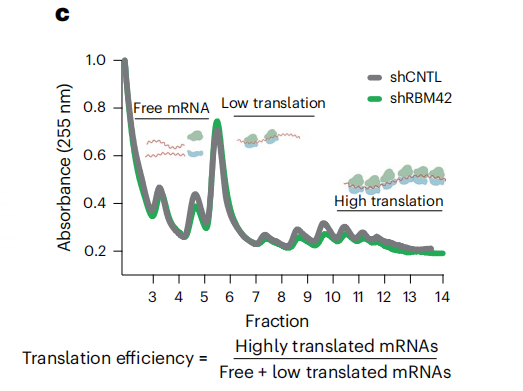
通过Polysome profiling计算TE[2]
这个技术原理是不是很复杂?
原理极其优雅简单——“以重取胜”。
想象mRNA是一条生产流水线,核糖体是工人在上面装配蛋白。
工效低:流水线上只有一个工人(单核糖体/80S),甚至没工人(游离RNA)。
工效高:流水线上挤满了工人(多聚核糖体/Polysome)。
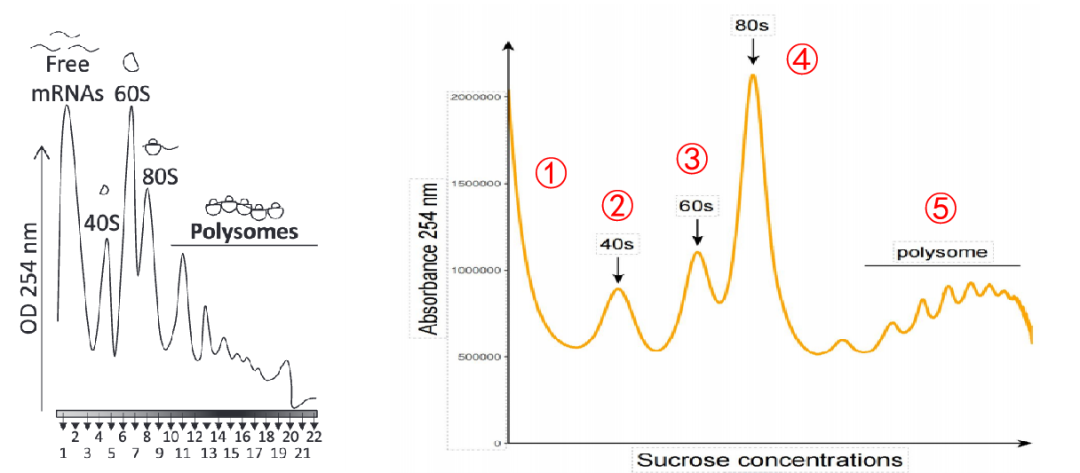
根据“结合核糖体越多,复合物越重”的物理特性,利用蔗糖密度梯度超速离心,就能把它们分离开。通过分析RNA到底是在“单人区”还是“多人区”,就能精准判断基因的翻译活性。
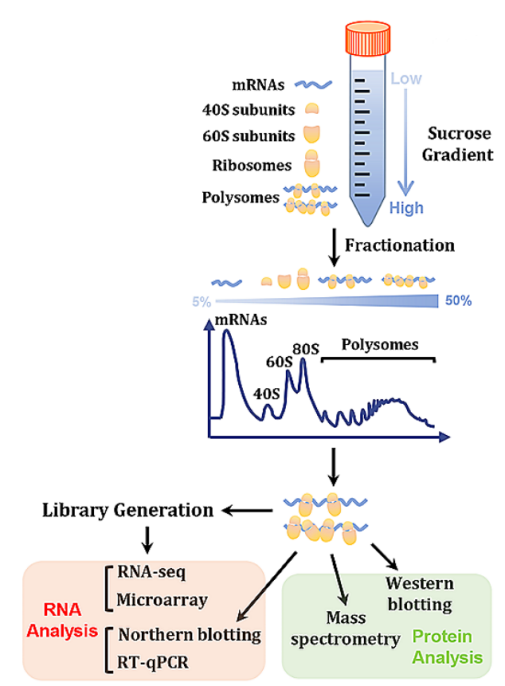
Polysome profiling技术路线图[1]
听说这实验很简单,自己实验室就能做?
原理懂了,实操却是“劝退级”难度。
很多同学觉得:“不就是配个蔗糖溶液离心吗?”
错!但不全错。
这个实验的门槛在于“又大又细”:
设备门槛高:你需要大型超速离心机(连续转数小时)和专用的密度梯度分馏系统(含UV检测和流量泵),一般实验室很难配齐。
操作容错低:梯度溶液配置繁琐,且必须在裂解液中加入放线菌酮(CHX)“锁住”核糖体。一旦操作不当,多聚核糖体解聚,跑出来的图谱就是一堆杂乱的峰,数据直接报废。
既然这么难,有没有“开挂”的解决方案?
有!吉赛生物Polysome Profiling PLUS版。
为了解决传统实验分辨率低、操作难、数据粗的痛点,我们推出了升级版攻略,专治各种不服。
升级一:从“大概看”到“高清看”
传统方法可能只收3-5个组分。PLUS版根据沉降系数,将产物精细分离为18个组分!
好处:RT-qPCR的数据点更密集,折线图更平滑,能捕捉到翻译复合物极微小的迁移变化。
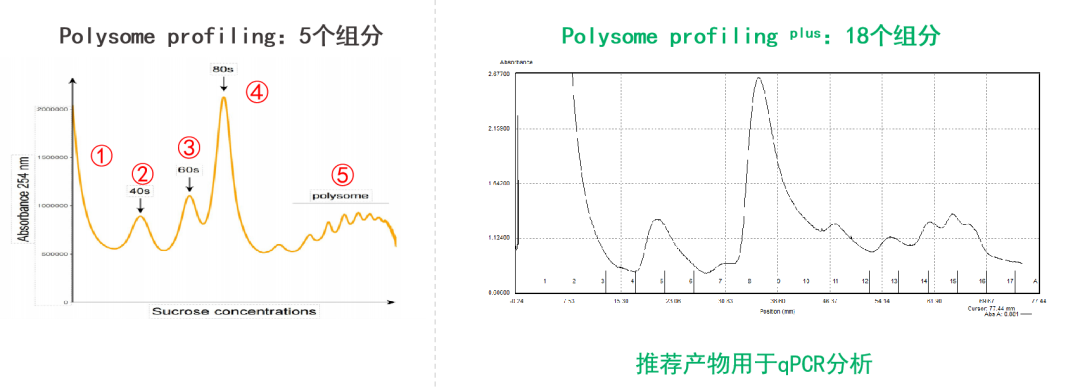
升级二:数据不仅要“全”,还要“准”
AUC统计:引入曲线下面积(AUC)分析,计算P/non-Pratio,用统计学数值量化样本的整体翻译活性。
严格质控:增加RNA完整性检测,根据质检结果调整上机量,确保下游测序不翻车。

升级三:打破样本“次元壁”
不仅能做细胞,连组织、细菌、真菌这种难搞的样本,PLUS版也能轻松搞定。
升级四:不仅仅是看编码基因
如果你在研究lncRNA或circRNA,PLUS版能帮你验证它们是否结合在多聚核糖体上。这是证明ncRNA具有翻译潜力(编码小肽)的硬核证据[3]!
小吉总结
如果你正苦恼于“有表型无机制”,或者“转录蛋白对不上”,千万别死磕RNA-seq了。
换个角度,从翻译组切入,利用Polysome Profiling PLUS拿到更精细的翻译效率数据,或许就是你文章冲刺高分的突破口!
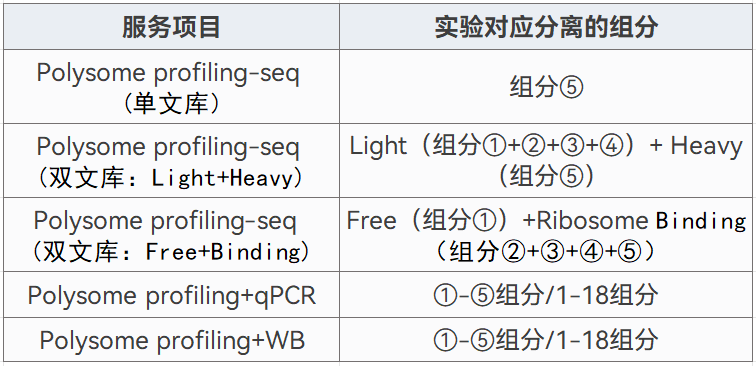
Polysome profiling 扩展包
想获取PLUS版详细技术资料?
在公众号后台回复关键词“Polysome”,即可查收样本前处理攻略及服务详情!
参考资料
[1] Su D, et al. Ribosome profiling: a powerful tool in oncological research. Biomark Res. 2024, 12(1):11.
[2] Kovalski JR, et al. Functional screen identifies RBM42 as a mediator of oncogenic mRNA translation specificity. Nat Cell Biol. 2025 Mar;27(3):518-529.
[3] Wu X, et al. A novel protein encoded by circular SMO RNA is essential for Hedgehog signaling activation and glioblastoma tumorigenicity. Genome Biol. 2021;22(1):33.

 购物车
购物车


 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn