
- 首页
- >
- GENESEED
- >
RNA pull-down探针法还是标签法?一文解决选择困难~
广泛应用于RNA-蛋白互作研究的技术,除了RIP,还有RIP的“CP”技术——RNA pull-down。
RIP是从已知蛋白出发,研究RNA的结合,针对目标蛋白下拉RNA;而RNA pull-down则是从已知的RNA出发,研究RNA结合蛋白(RBP),针对目标RNA下拉蛋白。两者互为补充、相互验证。
RNA pull-down技术主要有探针法和标签法两种,那这两种方法有什么区别呢?这里帮大家捋一捋~
RNA pull-down探针法
探针法原理(步骤)
① 制备探针:通过体外转录或化学合成制备生物素标记RNA探针,以模拟目标RNA分子。
② 裂解细胞:处理样本,得到细胞裂解液。
③ 磁珠偶联探针:利用链霉亲和素磁珠特异性捕获探针;
④ 捕获RBP:将磁珠-探针与细胞裂解液孵育,使探针捕获相关的RBP。
⑤ 分析产物:纯化产物,进行WB或质谱分析。
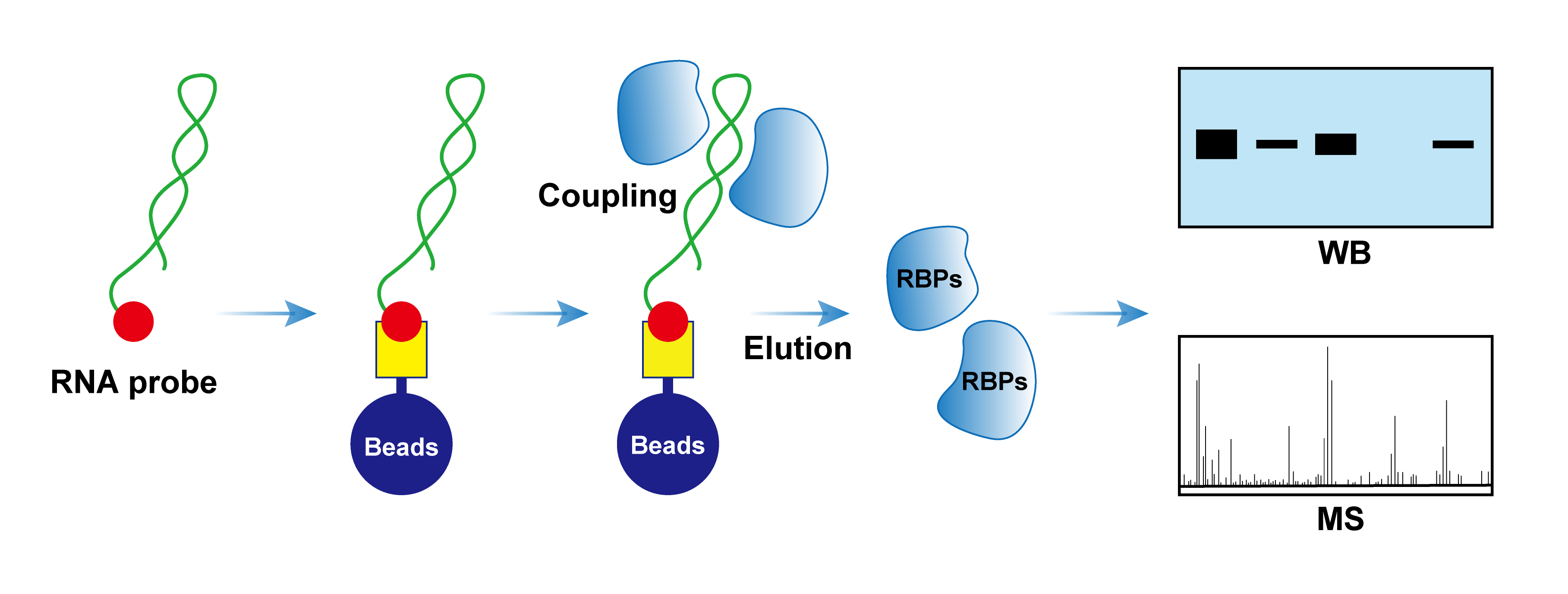
图1 RNA pull-down探针法原理
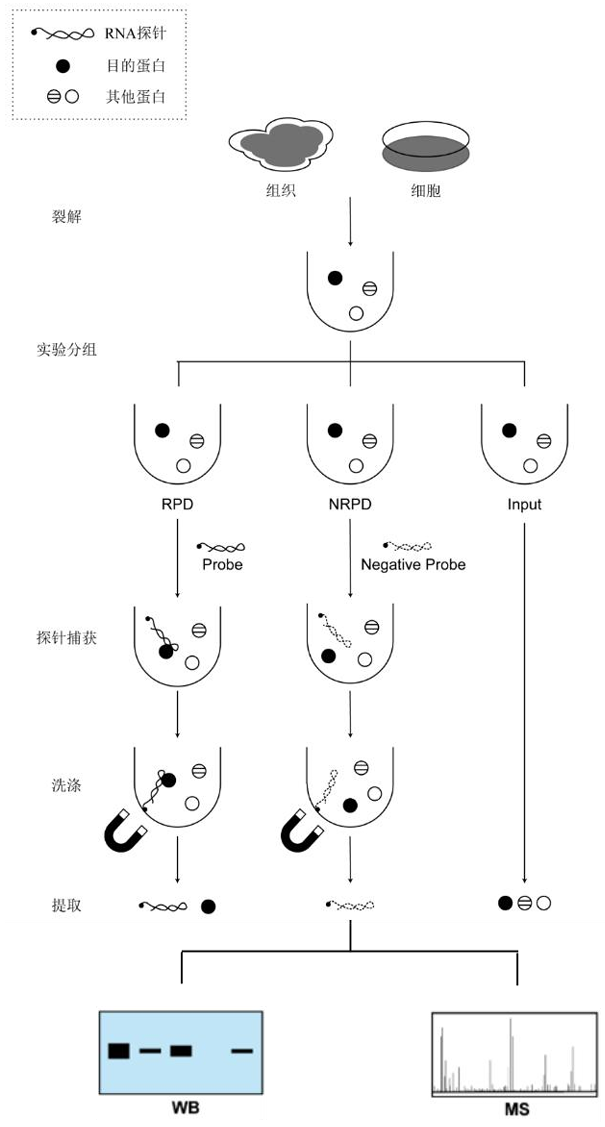
图2 RNA pull-down探针法实验流程
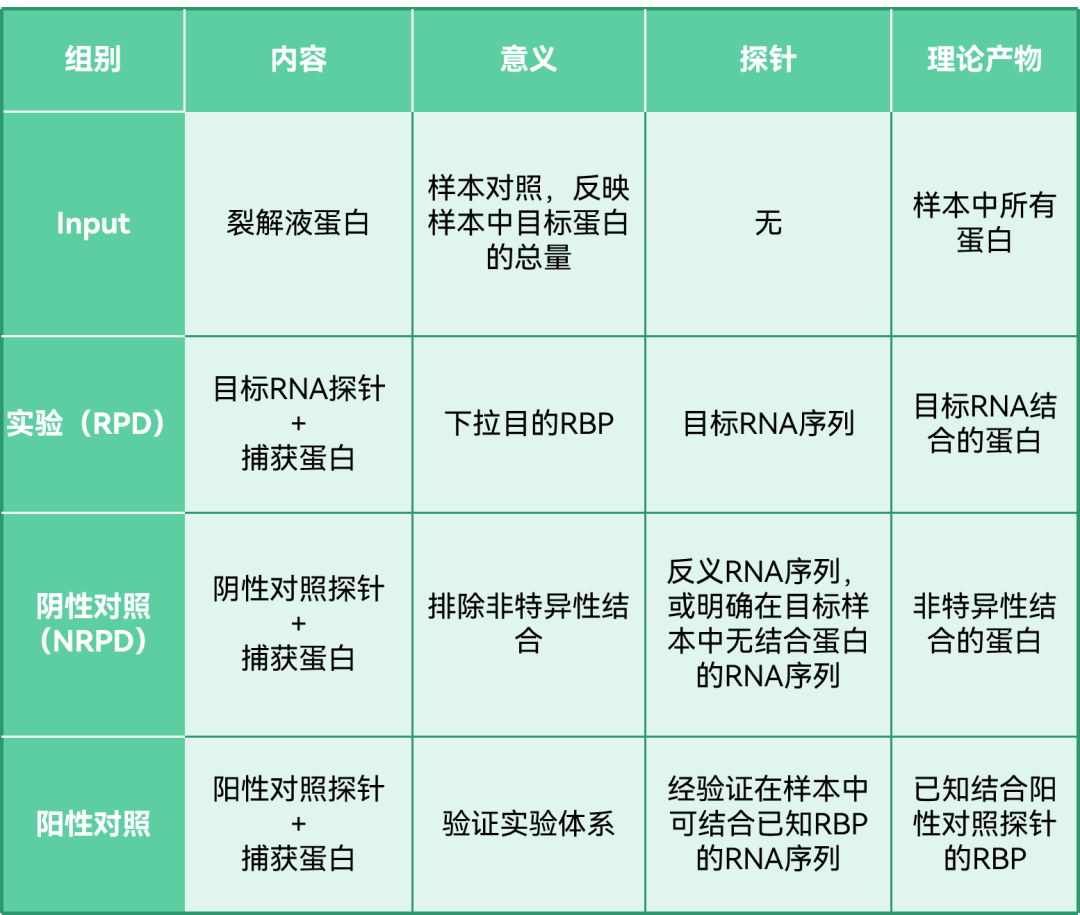
探针法实验分组
吉赛RNA pull-down探针法试剂盒操作视频

探针法优势
① 操作便捷:利用探针直接捕获细胞裂解液中的RBP,实验流程简单,效率高;
② 条件灵活可控:体外实验,影响因素和实验条件可控,实验风险低。
③ 探针设计灵活性高:可采用序列全长探针,也可设计截短或突变探针,研究RBP的具体结合片段或位点;不仅限线性的lncRNA和mRNA,还可通过探针序列设计应用于circRNA;
④ 适用范围广:不依赖于目标RNA本底表达量,对细胞内表达丰度低的RNA同样适用。
探针法应用案例
论文:circCDK13-loaded small extracellular vesicles accelerate healing in preclinical diabetic wound models
影响因子:15.7
研究内容:负载circCDK13的小细胞外囊泡促进临床前糖尿病创面愈合
为阐明circCDK13促进人皮肤成纤维细胞(HDFS)和人表皮角质形成细胞(HEKS)增殖和迁移的分子机制,研究进行了RNA pull-down(探针法)及银染和LC-MS/MS分析,实验结果联合catRAPID omics v2.0预测数据,筛选出IGF2BP3是可能与circCDK13相互作用的候选特异性RBP(图3b-d)。
进一步利用RNAfold(图3e)预测了具有最小自由能(MFE)的circCDK13二级结构;NPDock预测circCDK13和IGF2BP3的物理相互作用(图3f)。然后再进行RNA pull-down(探针法)-WB和RIP-qPCR,证实circCDK13与IGF2BP3之间的结合(图3g−i)。随后对预测的circCDK13结合位点和IGF2BP3结构域进行突变,进一步利用RIP和RNA pull-down技术分析了两者互作位点(图3k-o)。
研究应用了吉赛生物提供的RNA pull-down(探针法)试剂盒及RIP试剂盒。
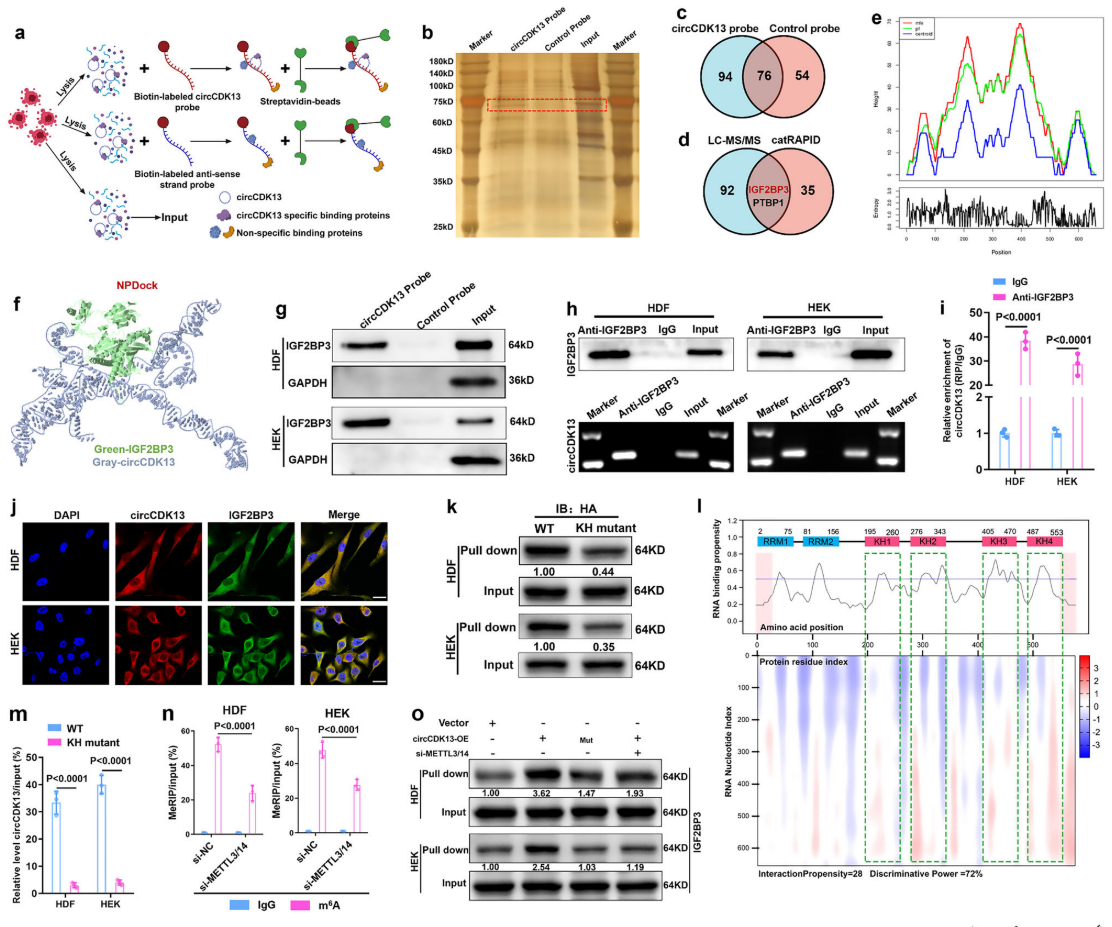
图3 a:RNA pull-down试验示意图;b:RNA pull-down产物银染;c:RNA pull-down产物LC-MS/MS分析结果;d:LC-MS/MS分析与catRAPID omics v2.0预测韦恩图;e:CDK13的最小自由能(MFE,红色)、热力学系综(绿色)和结构质心(蓝色);f:NPDock图形表示circCDK13和IGF2BP3蛋白的分子对接;g:RNA pull-down-WB实验证实circCDK13与IGF2BP3互作;h-i:RIP实验证实circCDK13与IGF2BP3互作;j:FISH和IF显示CDK13(红色)和IGFB2P3(绿色)共定位;k:RNA pull-dwon-WB证实IGF2BP3的KH结构域与CDK13结合;l:CatPAPID算法预测circCKD13-IGF2BP3互作功能结构域;m:RIP分析KH结构域突变对circCDK13富集度的影响;n:MeRIP分析METTL3/14干扰对CDK13的m6A修饰的影响;o:RNA pull-down-WB检测CDK13突变及METTL3/14干扰对IGF2BP3互作的影响。
RNA pull-down标签法
标签法原理(步骤)
RNA pull-down标签法原理基于MS2-CP系统。MS2适配体源自MS2噬菌体,是一段可形成发卡结构的RNA,可与噬菌体MS2外壳蛋白(CP)结合,且结合具有高度特异性和亲和力。
① 构建MS2-RNA载体:以MS2适配体RNA序列作为标签,插入到目标RNA中合适的位置,根据MS2-目标RNA序列构建表达载体;
② 构建CP蛋白载体:构建CP蛋白(带Flag标签)表达载体;
③ 共转染载体:将MS2-RNA表达载体和CP蛋白表达载体共转染到细胞中;
④ 形成复合物:在细胞中,MS2-目标RNA和CP蛋白表达后,目标RNA自然结合相应的RBP和RNA(如miRNA),CP-Flag蛋白结合MS2适配体。
⑤ 捕获复合物:然后裂解细胞,利用Flag抗体下拉CP-Flag-MS2-目标RNA-RBP/RNA复合物,对产物进行提取和纯化,即可得到目标RNA结合的RBP和RNA。
⑥ 分析产物:可分别对蛋白产物可进行WB、质谱;RNA产物可进行RT-qPCR、高通量测序分析。
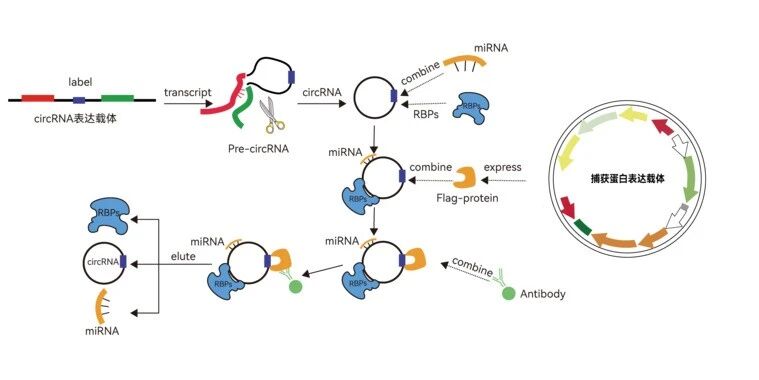
图4 RNA pull-down标签法原理(以circRNA为例)
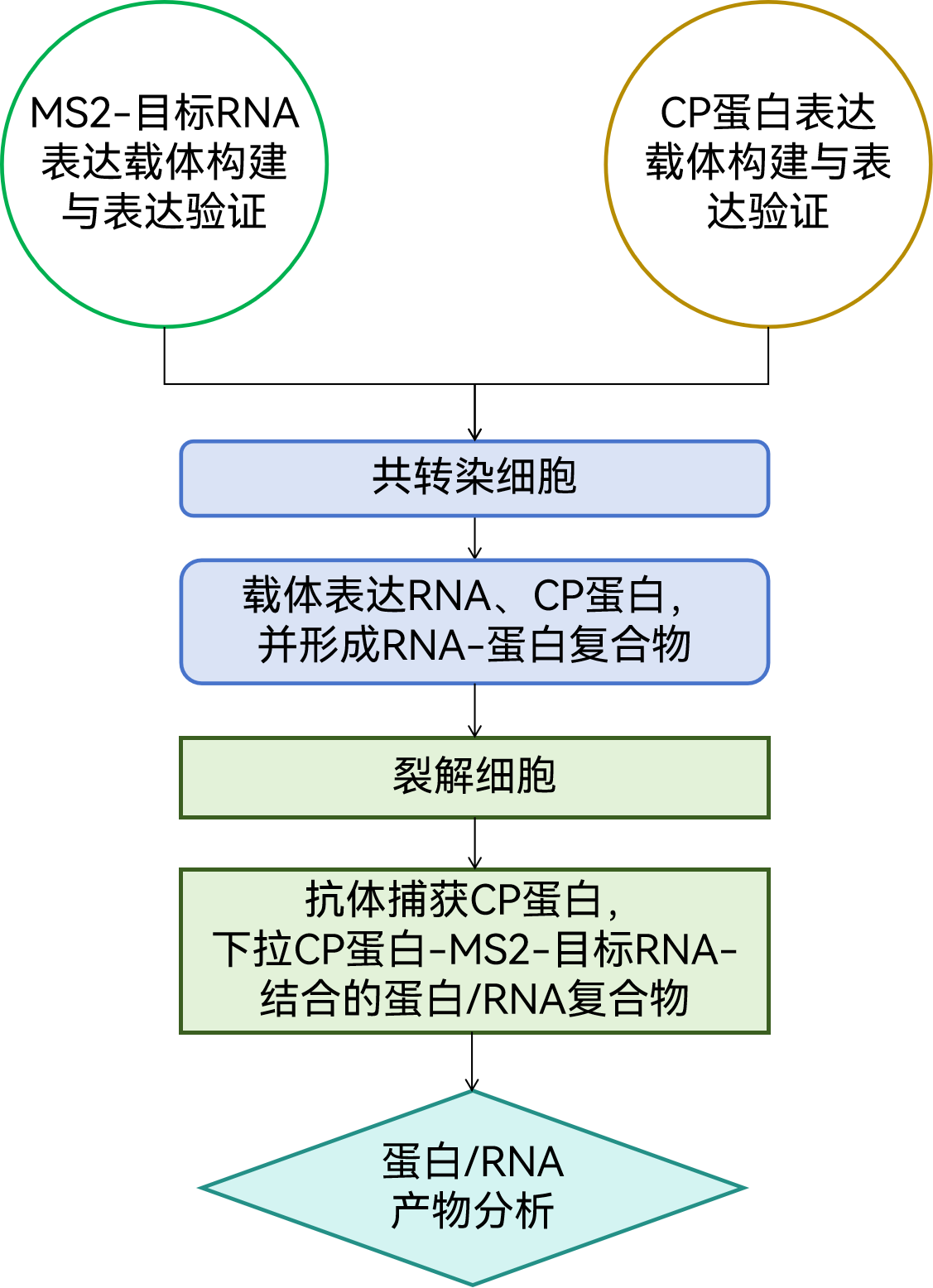
图5 RNA pull-down标签法操作流程
标签法优势
① 内源结合状态:RNA-蛋白复合物在细胞内生理状态下形成,更能反映真实的互作状态;允许对细胞进行不同处理,研究在不同条件下,目标RNA和蛋白或RNA的结合状态变化;
② 特异性更高:蛋白与RNA在体内的互作亲和力和特异性可能强于体外探针;
③ 适用范围广:可同时研究目标RNA结合的蛋白和RNA;不依赖于目标RNA本底表达量,对细胞内表达丰度低的RNA同样适用。
标签法应用案例
论文:Exosome-derived circUPF2 enhances resistance to targeted therapy by redeploying ferroptosis sensitivity in hepatocellular carcinoma
影响因子:12.6
研究内容:外泌体来源的circUPF2通过重新调控铁肝细胞癌的死亡敏感性,增强对靶向治疗的耐药性
研究利用RNA pull-down(标签法)和质谱分析鉴定了circUPF2的RBP,并进一步用RIP、RNA pull-down(标签法)-WB/qPCR等实验验证circUPF2、IGF2BP2蛋白和SLC7A11 mRNA之间的相互作用。
研究应用了吉赛生物提供的RNA pull-down(探针法)载体及质谱分析服务。
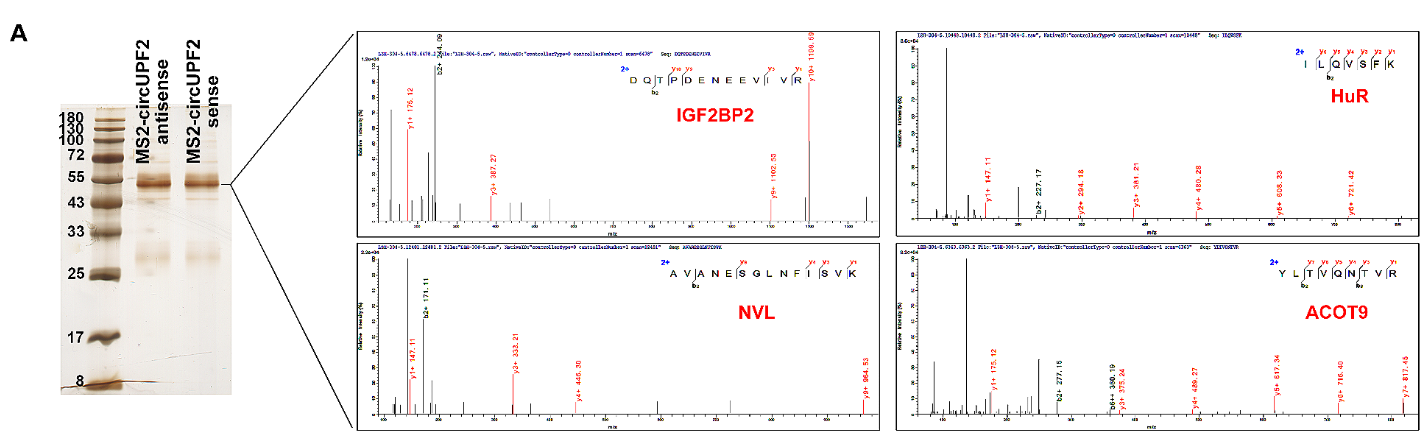
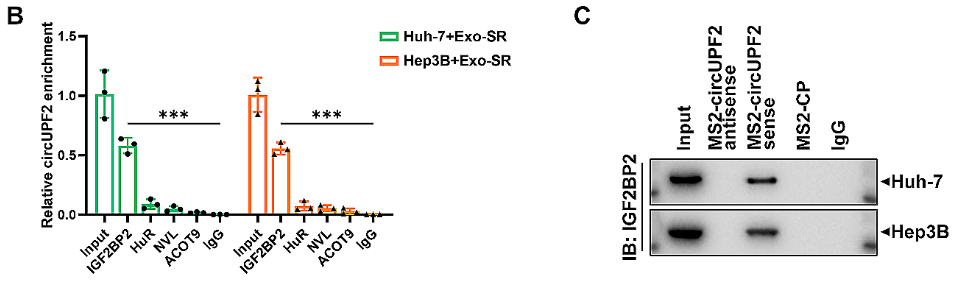
图6 A:RNA pull-down(标签法)联合质谱分析筛选肝癌细胞中circUPF2的RBP。B:RIP证实IGF2BP2是circUPF2的RBP。C:RNA pull-down-WB进一步证实circUPF2和IGF2BP2互作。
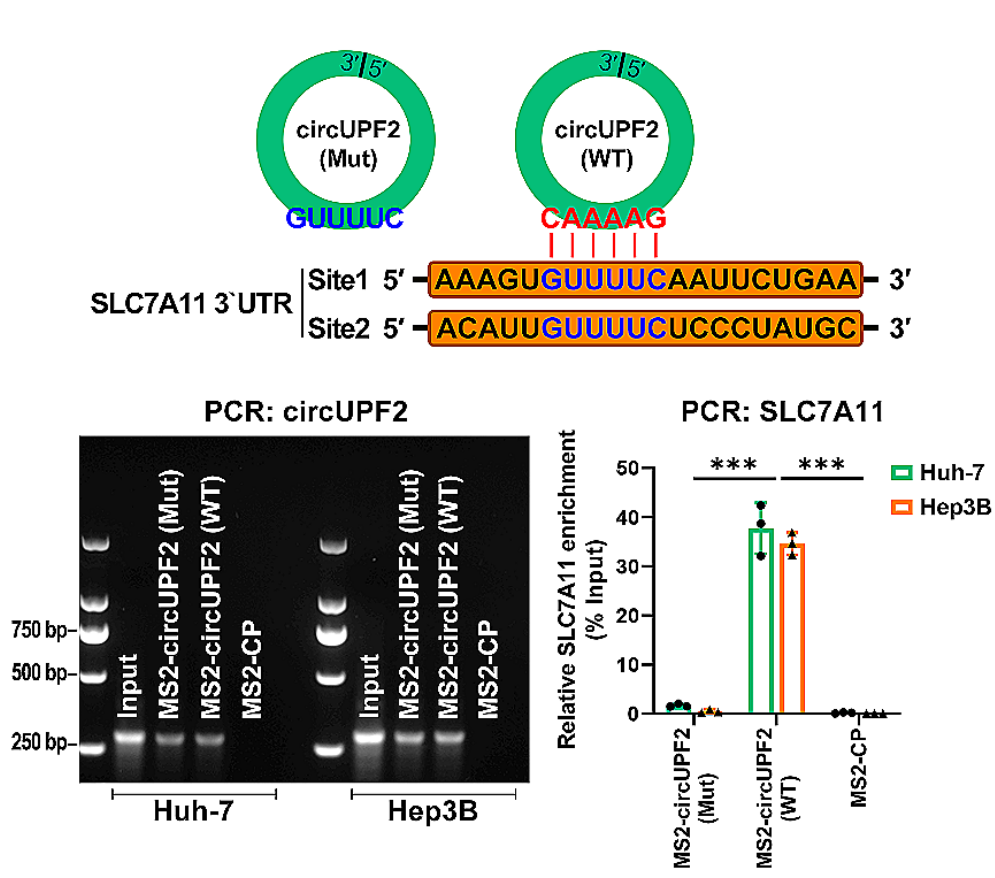
图7 Sequence BLAST分析表明,circUPF2直接靶向具有较高AU含量的SLC7A11 mRNA的3’UTR。RNA pull-down检测circUPF2(Mut)和circUPF2(Wt)的MS2-CP pull-down效率,并用qRT-PCR检测MS2-circUPF2富集SLC7A11 mRNA的相对丰度。
两种RNA pull-down对比汇总
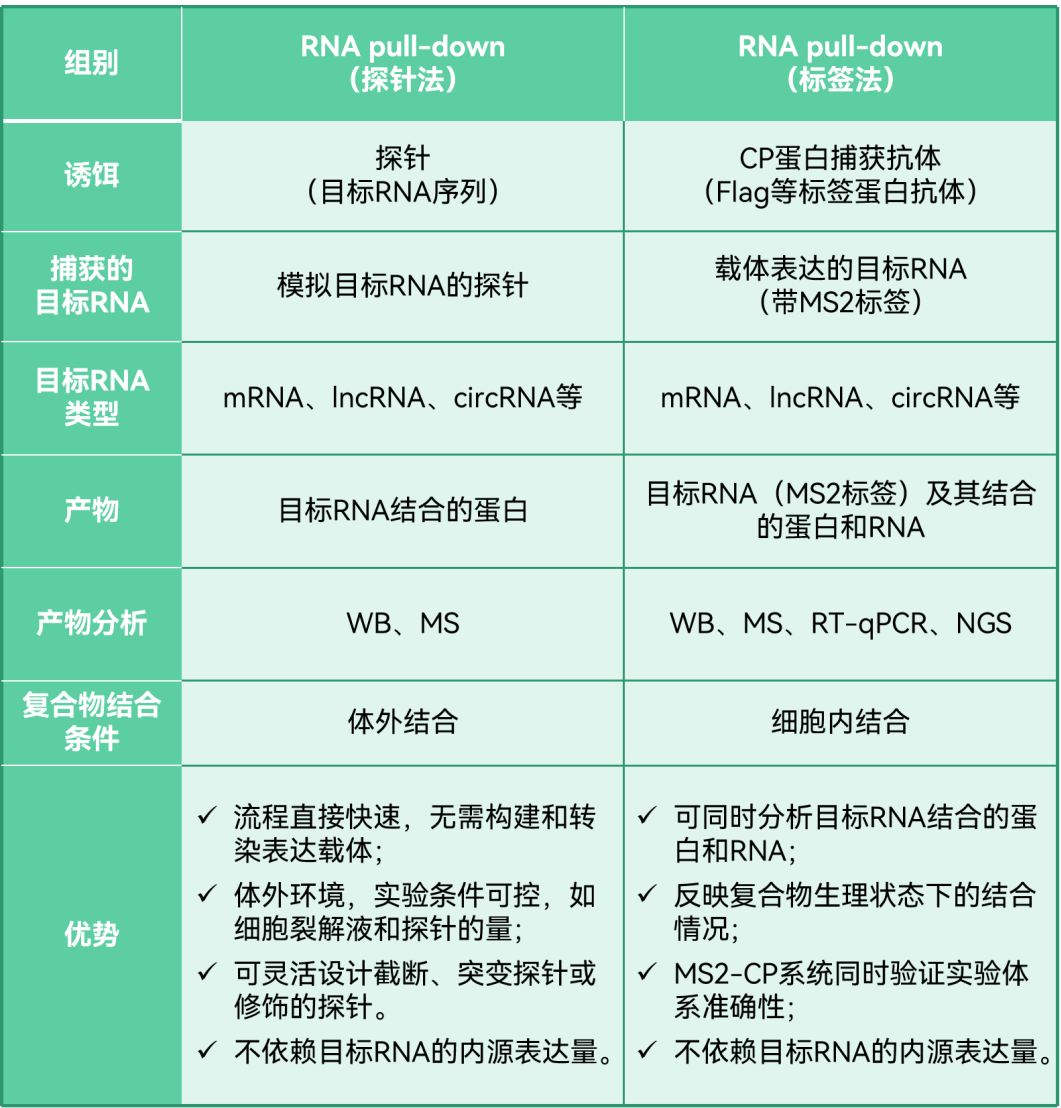
总之,探针法流程简便快捷,成功率高,对“新手”友好;而标签法步骤较复杂、操作难度较大,但可以反映复合物天然结合状态,还可以分析蛋白和RNA。
无论您选择RNA pulldown探针法还是标签法,吉赛生物都可以提供相应的产品或服务,全面满足您的科研需求!
吉赛生物RNA pull-down相关产品
技术 | 产品名称 | 产品货号 | 规格 |
生物素标记RNA探针制备 | GENESEED®T7 Biotin Labeled RNA Synthesis Kit | R0402 | 3 rxns |
RNA pull-down 探针法 | PureBinding®RNA-Protein pull-down Kit | P0201 | 12 rxns |
P0202 | 24 rxns |
吉赛生物分子互作解决方案
过表达与干扰 | ||
circRNA体外合成 | mRNA体外合成 | LNP包封 |
过表达载体 | shRNA载体 | 病毒包装 |
siRNA、ASO | miRNA mimic、miRNA inhibitor | Agomir、Antagomir |
分子共定位 | ||
FISH | IF | IF-FISH |
分子互作分析 | ||
蛋白-DNA互作 | ChIP、DNA pulldown | |
RNA-DNA互作 | ChIRP | |
RNA-RNA互作 | RNA pull-down(标签法)、RAP | |
RNA-蛋白互作 | RIP、CLIP、RNA pull-down | |
蛋白-蛋白互作 | Co-IP | |
分子互作验证 | 荧光素酶报告检测 | |

 购物车
购物车


 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn